线粒体测序
线粒体测序简介
线粒体疾病是一种异质性疾病,可能发生在生命的任何阶段。由于个体间表型的可变性和遗传异质性,线粒体疾病相关突变的研究仍旧存在挑战。
利用新一代测序(NGS)技术进行线粒体测序可以解决这些问题,该方法能全面检测和分析线粒体疾病相关变异。NGS还能将线粒体DNA分析用于其他应用,例如人类身份鉴定、法医和癌症研究。
基于NGS的线粒体测序的优势
NGS线粒体测序可实现:
- 检测常见和罕见的线粒体点突变与缺失1,2,3,4
- 线粒体DNA(mtDNA)和核DNA(nDNA)的快速分析2,4
- 准确、灵敏地测量异质性1(给定的样本中包含多个线粒体DNA序列变异)
- D环高度变异区域(一种非编码的mtDNA区域,是遗传变异研究的热点区域5)和/或整个线粒体基因组的分析
线粒体测序研究解决方案
常见的线粒体疾病相关突变NGS分析方法包括全外显子组测序和靶向基因测序。
全外显子组测序能分析基因组的蛋白编码区域,如果有需要,还可以添加非翻译区(UTR)和microRNA。
靶向基因测序重点关注具有已知或疑似疾病关联的特定基因或目标基因区域。
比较下表中的两个方法。

比较线粒体测序方法
| 方法 | 全外显子组测序 | 靶向测序 |
| 说明 | 提供线粒体基因组编码区的完整视图。能让研究人员更深入地观察经常出现致病性变异的线粒体区域。 | 专注于特定的线粒体基因或区域。利用基于线粒体疾病相关变异的先验知识选择的测序panel。 |
| 优点 |
|
|
| 考虑因素 |
|
|
| 更多信息 | 深入了解外显子组测序 | 深入了解靶向测序 |
分析mtDNA、piRNA和T细胞受体库的多样性
西奈山的研究人员利用Illumina测序技术开发出了新方法,可用于研究线粒体DNA、piwi相互作用RNA和T细胞受体库的多样性。他们的目标是揭示癌症、不孕不育和自体免疫性疾病的机制。
阅读文章
特色mtDNA测序工作流程
采用这种简单、集成的工作流程分析线粒体疾病相关变异。
数据分析
BaseSpace mtDNA Variant Processor App
可简化D环和完整线粒体DNA测序数据的变异分析。
BaseSpace mtDNA Variant Analyzer App
可简化D环和完整线粒体DNA测序数据的可视化处理。

使用mtDNA分析来进行法医学和人类身份鉴定研究
由于每个细胞中存在成百上千个mtDNA拷贝,因此它可以在许多核DNA不能保存的环境中保存。这让mtDNA分析成为人类身份鉴定的强大工具。然而,异质性(mtDNA序列可能具有组织类型特异性,甚至在单个细胞或组织类型中都可能有所变化)会使法医学鉴定所需的样本关联性确认工作变得困难。
Illumina NGS能够实现线粒体DNA异质性的准确检测和混合样本的高分辨率。
用深度mtDNA测序分析骨骼残骸
法医学研究人员使用Illumina NGS对mtDNA的高度变异区进行测序来协助骨骼残骸鉴定。
更多资源
参考文献
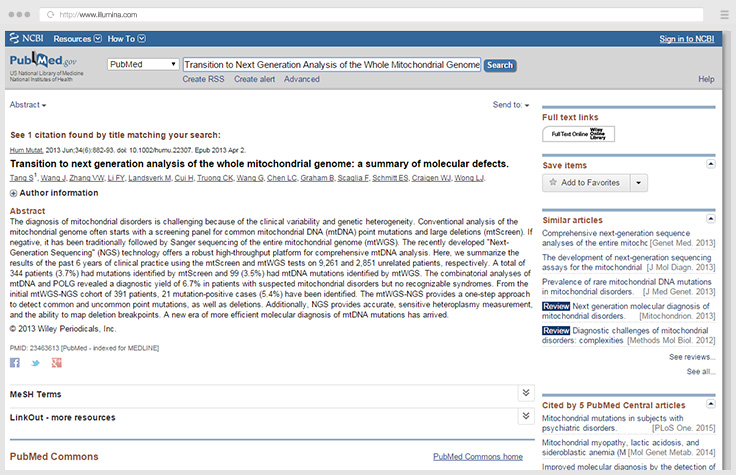
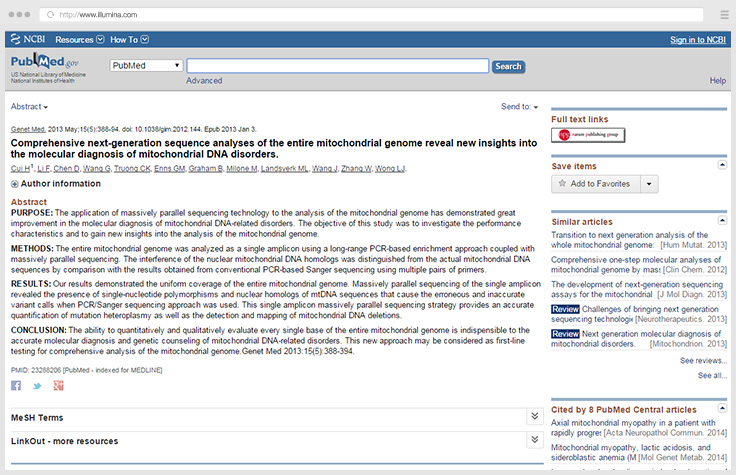
- Tang S, Wang J, Zhang VW, et al.Transition to next generation analysis of the whole mitochondrial genome: a summary of molecular defects.Hum Mut.2013;34:882-893.
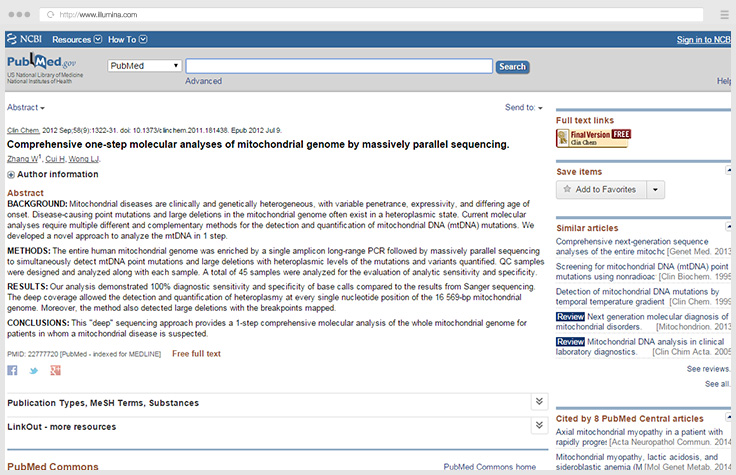
- Wong LC.Next generation molecular diagnosis of mitochondrial disorders.Mitochondrion.2013;13:379-387.
- Chinnery PF, Elliott HR, Hudson G, Samuels DC, Relton CL.Epigenetics, epidemiology and mitochondrial DNA diseases.Int J Epidemiol.2012;41:177-187.
- Calvo SE, Compton AG, Hershman SG, et al.Molecular diagnosis of infantile mitochondrial disease with targeted next-generation sequencing.Sci Transl Med.2012;4:118ra10.
- Sharma H, Singh A, Sharma C, Kumar Jain S, Singh N. Mutations in the mitochondrial DNA D-loop region are frequent in cervical cancer. Cancer Cell Int. 2005;5:34.